2020年5月,苏州系统医学研究所李贵东课题组、美国加州理工学院戴维·巴尔的摩课题组、美国西雅图系统医学研究所詹姆斯·希斯课题组在Nat Communication(IF:12.121)上发表“多mic单细胞快照揭示了黑色素瘤细胞系中多个独立的“耐药T细胞”,通过单细胞蛋白质组学和单细胞代谢组学,揭示了黑色素瘤细胞在药物反应和耐药性之间的早期变化轨迹。发现初始药物反应状态和药物耐受状态之间存在两条完全不同的途径,这一发现得到了实验和理论的证实。研究结果极大地加深了我们对肿瘤细胞耐药性转化的认识,也为今后设计更有效的肿瘤联合治疗提供了新的指导。

中文标题:单细胞多显微术揭示黑色素瘤细胞系中多种独立的耐药途径
对象:黑色素瘤细胞
出版期刊:《国家通讯》
影响因素:12.121
发表日期:2020年5月
生物技术应用:单细胞蛋白质组学、单细胞代谢组学
研究背景通过细胞的高维状态来确定单细胞的变化轨迹,是理解感染细胞在药物作用下从细胞分化到表观遗传学的生物学变化的巨大挑战。通过数据预测和实验,作者确定了BRAFV600E突变的黑色素瘤单细胞从药物敏感状态到药物耐受状态的轨迹。虽然单细胞组学可以获得细胞在某一时刻的状态,但随机的细胞状态切换可能会混淆单细胞变化的轨迹。作者分析了药物治疗5天内的信号通路、表型和代谢调节因子,发现连接药物敏感性和药物耐受性的通路有两条,每条通路都表现出独特的药物敏感性,更新了等基因细胞群中耐药的发展模式。
研究思路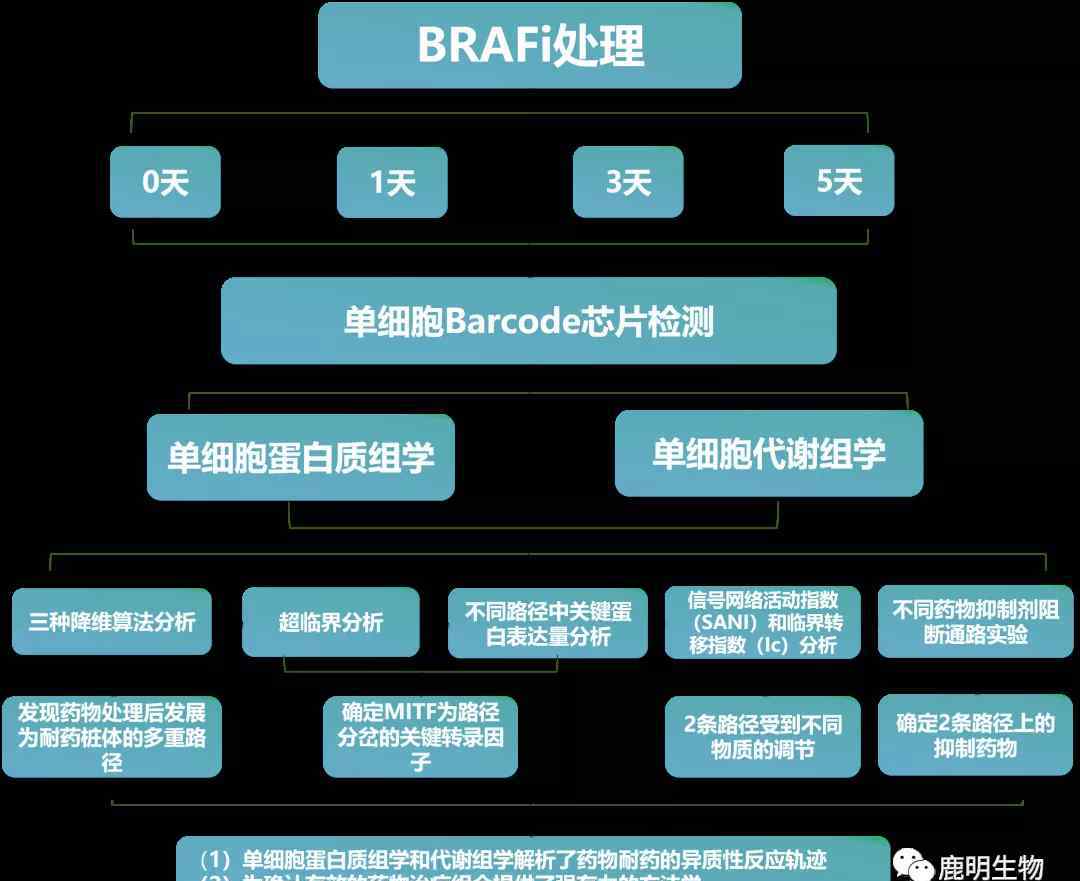
1.单细胞蛋白质组学和单细胞代谢组学分析。BRAFi治疗
单细胞条形码芯片(SCBC)用于检测在BRARi中培养5天的BRAFV600E的突变细胞M397的蛋白质(定制的蛋白质芯片)和葡萄糖,以确定单个黑色素瘤细胞的耐药性(图1a)。
药物治疗0、1、3和5天后(D0为对照组,D1、D3和d 5为实验组),将单细胞分离到SCBC的纳升微室中。每个分离的细胞被原位裂解,从而释放其细胞内容物。SCBC的每个微室都包含一个完整的条形码阵列,每个条形码要么是蛋白质的特异性抗体,要么是检测特定代谢物的分子探针(图1a)。研究人员收集了各种癌细胞水平的蛋白质和代谢标记,用于定制单细胞条形码芯片。
与D0天药物治疗相比,不同天数药物治疗后,部分检测物质在单细胞水平上有较高的变化(图1b,c)。例如黑色素瘤细胞的转录因子MITF、代谢调节因子HIF1α和p-AMPKα、增殖标记物Ki67、葡萄糖等。在药物治疗的初始阶段(D1),葡萄糖摄入、大多数代谢调节因子、磷酸化蛋白和Ki67被抑制,这反映了BRAFi对关键致癌信号通路的抑制作用。(在药物治疗的其他日子里,也有被检测物质的具体变化。)
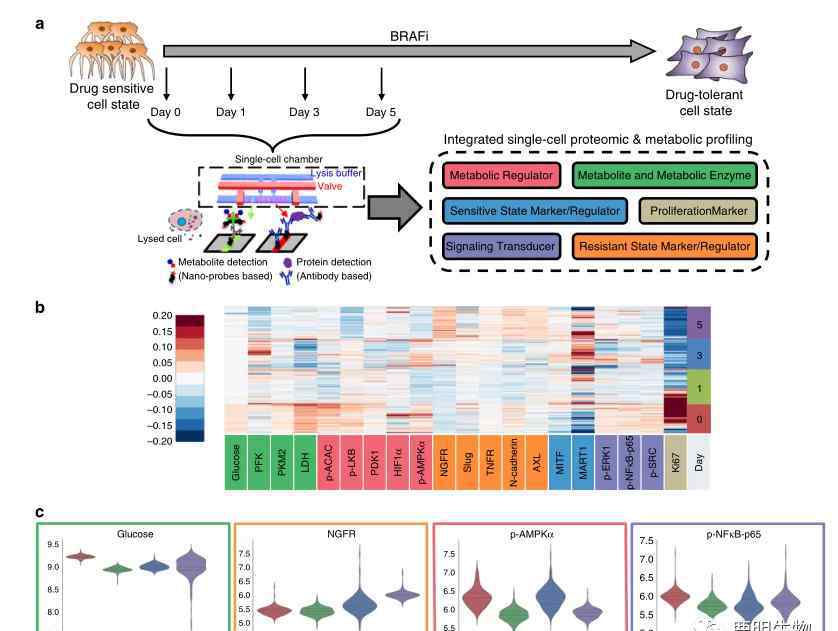
图1 |不同时间段药物处理的M397细胞单细胞蛋白质组学和单细胞代谢组学分析
单细胞蛋白质组学和代谢组学联合分析的实验设计。单细胞条形码芯片(SCBC)技术用于采集和分析BRAFi处理过程中不同处理时间点细胞的蛋白质组和代谢产物。每个细胞有6个不同的标记。
蛋白质组学和代谢组学数据的聚类热图,其中行代表具有不同治疗持续时间的药物的单细胞样本,列代表检测到的物质,不同的颜色代表检测到的物质的测量丰度水平。最后一列表示BRAFi治疗的天数。X轴上被检测物体的颜色对应其所属的六种不同标记;
(c)小提琴图显示了在四个时间点检测到的一些代表性物质的丰度。y轴代表被测物质丰度值的对数值。
2.降维分析发现药物治疗后从药物敏感状态到药物耐药状态有多条路径
用三种降维算法(FLOW-MAP47、t-SNE48和PHATE49)在单细胞水平上观察了不同时间段BRAFi处理后几种物质的变化。视觉图显示黑色素瘤细胞根据药物治疗的持续时间聚集(图2),显示除细胞状态之外的时间轨迹。细胞耐药性在第0天和第1 /3天至第5天的分叉也表明了从药物敏感状态到药物耐药状态的两条途径的可能性。
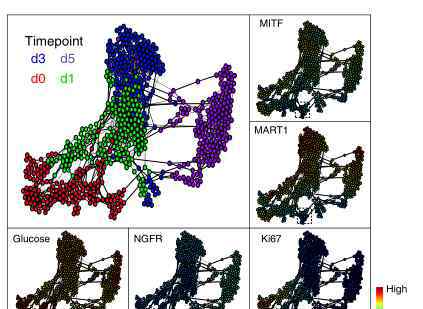
图2 |单细胞蛋白质组和单细胞代谢组学整合数据的可视化
每个点代表一个单元格。每对细胞之间的距离代表了它们之间蛋白质组和代谢物的总体差异。单个单元格之间有一条线足够近。左上角方框中不同的颜色表示BRAFi处理细胞的时间(0、1、3、5天)。其他方框中点的颜色表示单细胞中每种检测物质的丰度。MITF、MART1和Ki67中的虚线框代表第0天的细胞亚群,其中三种物质的丰度较低。
3.令人惊讶的分析揭示了不同路径中被检测对象的聚合模块
为了进一步分析耐药性的分子轨迹,对该单细胞数据集进行了惊奇分析,确定了两个主要模块,每个模块代表一组检测物质。单个单元中模块的影响分数表示单元中与模块相关的分析物的富集或抑制程度,并通过对流程图中每个节点的模块1和2的影响分数进行颜色编码而可视化(图3a)。作者发现,模块1在上下路径上的影响评分由负(蓝色)逐渐增加到正(红色),在中间时间点有明显的正负变化(图3a-左)。当模块2的影响分数投射到流程图上时,有相同的变化,这表明有一个生物物理屏障将上下路径分开(图3a-右)。
值得注意的是,表型转录因子MITF及其下游蛋白MART1在黑色素瘤细胞中的表达与模块2评分呈负相关(图3b),这表明两条途径的分叉可能与这两种蛋白密切相关。

图3 |令人惊讶的分析表明,MITF是路径分叉的关键转录因子
(a)直观分析两个模块的影响得分。模块1依赖于时间,模块2显示了特定的路径。黑色虚线表示每个单个单元格对应的模块分值接近零的区域;
单细胞检测对象的丰度与模块2的得分之间的皮尔逊相关;
(c,d)在第0天,模块2中Ki67和MITF表达水平的框图被分为两个亚组:高和低。每组16个生物重复;
(e)在第0天,通过定量聚合酶链反应检测MITF高表达细胞和MITF低表达细胞中Ki67的表达,每组重复3次生物学操作;
(f)在治疗开始时,在0天组的MITF高表达细胞和MITF低表达细胞中测量倍增时间。每组3次生物重复;
(g)用BRAFi处理5天的细胞通过单细胞延时显微镜进行分析。
以上四张图:BRAFi治疗5天后GFP低表达和GFP高表达的延迟图像。三次生物重复。
下图:单细胞MITF随时间的变化,MITF高表达(橙色),MITF低表达(蓝色)。
粗线代表平均值;
(h)分析第5天收集的单细胞数据。模块2中蛞蝓、MITF、MART1和PFK的相对表达水平分为高和低两个亚组,每组16个生物重复。
(1)在BRAFi处理5天后,在MITF-高表达细胞和MITF-低表达-0天细胞中使用qPRC检测蛞蝓、MITF、Mart1和PFK的表达水平,每组3次生物重复。
4.路径分叉的实验验证
惊奇分析预测从药物敏感状态到药物耐受状态有两条路径。然后,需要实验来验证单个单元是沿着单个轨道在两种状态之间切换,还是在多条路径之间随机切换。实验结果显示,与MITF低表达亚群相比,MITF高表达细胞显示出更高水平的Ki67和MITF丰度,并且具有更短的倍增时间(图3c-f)。
为了量化药物治疗期间MITF高表达细胞和MITF低表达细胞之间随机相互转化的频率,作者在BRAFi治疗后5天内在大量单细胞中检测到MITF活性。正如预期的那样,MITF高表达细胞中的MITF活性显示出比MITF低表达细胞中的活性更高(图3g),并且没有观察到两个基因座之间明显的随机转换。
为了进一步确认给药5天后分类细胞的状态,作者定量了给药5天后上和下通路之间差异表达的检测物质。单细胞数据集的挖掘解释了许多检测到的物质,包括蛞蝓、MITF、MART1和PFK,在给药5天后在两条途径(模块2的正值和负值)之间差异表达(图3h)。
通过分析药物治疗5天后MITF-高表达细胞和MITF-低表达-第0天细胞中Slug、MITF、MART1和PFK的表达,作者发现MITF-低表达细胞中四种基因的表达水平在治疗5天后显著低于MITF-高表达细胞中的表达水平(图3i)。
5.临界点分析决定了轨迹分叉的调节物质
对单个细胞数据集的意外分析表明,在D1-D3时间窗(图3,左)中,上下通路都具有细胞状态转换的特征。
为了识别上下路径上的每个临界点,作者首先将所有时间点的单细胞数据聚合成流程图上的14个不同的子群(图4a)。每个集群代表一个过渡中间状态。集群1、6、7、8、10、11和12位于上部路径,而集群2、3、9、13和14位于下部路径。
信号网络活动指数(SANI)和临界跃迁指数(Ic)用于预测高维状态下的临界跃迁。作者发现,位于上方路径的聚类7和聚类9在其各自的路径中具有最高的SANI值(图4b,c)。这表明集群7和集群9最接近这两条路径上的临界点。
通过对集群7和集群9临界点的网络分析,发现这两个集群的网络具有不同的结构特征(图4d,e),这意味着这两个集群以不同的方式调节细胞群的跃迁。网络中的中枢调节物质可能是潜在的药物靶点。在集群7(上部途径)中,发现一些转录因子和代谢酶,包括MITF、PFK、p-LKB、PKM2、LDH2和蛞蝓,在网络中显示出高连接性(图4f)。对于Cluter 9(下通道),TNFR、N-钙粘蛋白和p-NFκB-p65在网络中显示高连通性(图4f)。一个有趣的观察结果是,聚类7中得分高的物质在聚类9中得分低,反之亦然,这表明这两条路径受到不同的调节。

图4 |两个轨道的临界点分析和网络分析
所有细胞在四个时间点聚集成子集。左图颜色根据药物治疗的不同时间段进行编码。右边的颜色是根据聚类分析定义的14个子组编码的。
(b)上路径临界点的过渡分析。在与上行链路路径相关联的每个子组中计算临界点指数SNAI,并在图上用不同的颜色编码。红色表示SNAI值较高,而蓝色表示SNAI值较低。聚类7显示在上部路径中最高的SNAI值;
下路径的临界点转换分析。在与下行路径相关联的每个子组中计算临界点指数SNAI,并用不同的颜色在图上编码。红色表示SNAI值较高,而蓝色表示SNAI值较低。聚类9显示了较低路径中最高的SNAI值;
以及(d)从聚类7的SCBC提取数据,并分析物质-物质相关网络。相关的强度体现在每个圆圈的颜色上(橙色表示正相关,蓝色表示负相关);
(e)从第9类SCBC提取数据,并分析物质-物质关联网络。相关的强度体现在每个圆圈的颜色上(橙色表示正相关,蓝色表示负相关);
(f)每个网络中每个节点的重要性得分分析由节点的度(量化网络中每个节点的连通性)来定义。颜色表示集群7或集群9网络中每个节点的节点度值。高分节点假设为重要节点,标有星号的节点进一步被药物干扰。
6.确定干扰两条路径的物质
为了测试这两种途径的变化是否由不同的调节物质驱动,作者选择了两种特异性靶向PKM2或NF-κB的抑制剂分别干扰簇7和簇9。选择MITF高表达细胞和MITF低表达细胞亚群,用蛋白激酶2抑制剂或核因子κB抑制剂联合布拉菲治疗。根据作者的假设,MITF低表达细胞亚群对BRAFi+NF-κBi组合更敏感(图5a),而MITF高表达细胞亚群对BRAFi+PKM2i组合更敏感(图5b)。在MITF基因敲除的M398细胞系上检测到相同的药物组合,结果也验证了作者的假设(图5c,d)。因此,不同轨迹的细胞对PKM2和NF-κB的抑制表现出不同的敏感性。
考虑到这两条途径的调节依赖性的差异,作者进一步假设,通过同时抑制PKM2和NF-κB信号传导来阻断这两条途径可能在防止向BRAFi耐受性的转变中表现出相加效应。为了验证这一假设,作者用三种药物组合(BRAFi+PKM2i+NF-κBi)体外处理M397细胞5天,并比较了单种药物(仅BRAFi)和双种药物组合(BRAFi+PKM2i和BRAFi+NF-κBi)5天获得的细胞数。与作者的预测一致,三种药物组合明显优于两种药物组合,两种药物组合优于单一疗法(图5e)。这意味着这些药物可能通过选择性阻断BRAFi诱导的细胞状态向耐药状态的转变而发挥作用。这些结果表明上下通路是独立的,不同的调节物质和相应的药物可以阻断通路。
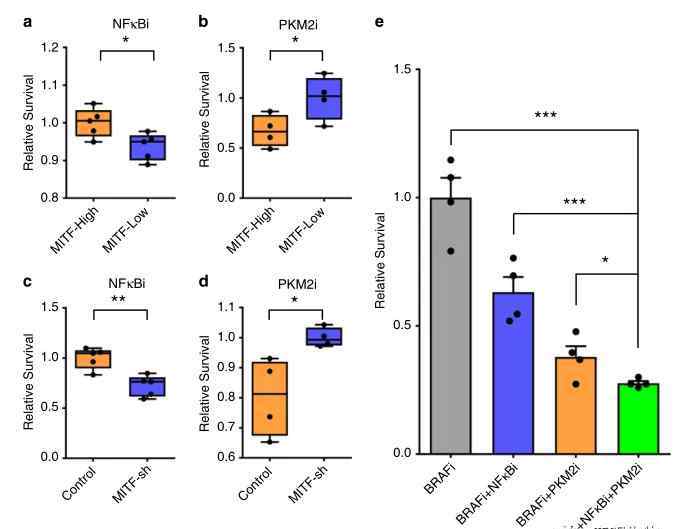
图5 |与细胞中两条轨迹相关的药物敏感性差异
(a)给药前对MITF-绿色荧光蛋白报告细胞系的MITF高表达和MITF低表达亚群进行分类。然后用BRAFi+NF-κBi处理5天,收集细胞计数。MITF高表达细胞和MITF低表达细胞的相对存活率在用BraFi+NF-κB联合治疗5天后绘制。存活数据根据MITF高表达样本进行标准化。每组4次生物重复;
和(b)在给药前对MITF-绿色荧光蛋白报告细胞系的MITF高表达亚群和MITF低表达亚群进行分类。然后用BRAFi+PKM2i处理5天,收集细胞计数。MITF高表达细胞和MITF低表达细胞的相对存活率在用BRAFi+PKM2i联合治疗5天后绘制。存活数据根据MITF低表达样本进行标准化。每组4次生物重复;
(c)用BRAFi+NF-κBi处理MITF敲除细胞和对照细胞5天,并收集细胞计数。在brafi+nfκb联合治疗5天后,计算对照组和MITF-sh细胞的相对存活率。生存数据根据对照组样本进行标准化。每个实验都是每组5个独立生物样本的结果。
(d)用BRAFi+PKM2i处理MITF敲除细胞和对照细胞5天,并收集细胞计数。绘制对照组和MITF-sh细胞经BRAFi+PKM2i联合治疗5天后的相对存活率。生存数据根据MITF sh样本进行标准化。每组4次生物重复;
(e)收集用BRAFi、brafi+nf κ bi、BRAFi+PKM2i和BRAFi+NFκBi+PKM2i处理5天的M397细胞并计数。绘制处理5天后细胞的相对存活率,包括BRAFi、BRAFi+NF-κBi、BRAFi+PKM2i或BRAFi+PKM2i+NF-κBi。生存数据根据BRAFi单一疗法治疗的样本进行标准化。每组有4个生物重复。
研究结论本研究通过单细胞蛋白质组学和代谢组学分析了耐药性的异质性反应轨迹,更新了目前对耐药性发展的认识,为确定有效的治疗组合提供了有力的方法。

李桂东教授介绍
李桂东研究员毕业于厦门大学,并于2012年在加州大学欧文分校获得博士学位。后来,他加入诺贝尔奖获得者戴维·巴尔的摩,在加州理工学院的实验室做博士后研究。课题组研究方向:建立TCR抗原靶筛选平台,筛选和识别潜在的新肿瘤治疗靶点;研究抗原特异性T细胞在感染、肿瘤和自身免疫性疾病中的反应;探讨肿瘤微环境对免疫疗法的耐药机制。
参考文献:多原子单细胞快照揭示黑色素瘤细胞株耐药性的多条独立轨迹[J]。《自然通讯》,2020年。
猜你还想看◆蛋白质组学:重症患者血清蛋白质组学和代谢生物标志物的细胞重新冠肺炎筛选
◆蛋白质组学:细胞热点|通过分馏质谱对植物蛋白质组的定量研究为遗传学和突变表型提供了框架
◆代谢组学:【小分子,大能量】自然评论|综述三种肠道细菌代谢产物在炎症性肠病中的作用
◆代谢组学:【胆汁酸系列】自然|胆汁酸次级代谢产物促进结肠组织Treg细胞的产生
结束
1.《单细胞蛋白 单细胞蛋白质组学和代谢组学揭示黑色素瘤耐药进化轨迹》援引自互联网,旨在传递更多网络信息知识,仅代表作者本人观点,与本网站无关,侵删请联系页脚下方联系方式。
2.《单细胞蛋白 单细胞蛋白质组学和代谢组学揭示黑色素瘤耐药进化轨迹》仅供读者参考,本网站未对该内容进行证实,对其原创性、真实性、完整性、及时性不作任何保证。
3.文章转载时请保留本站内容来源地址,https://www.lu-xu.com/shehui/1115215.html