拓扑关联域(TAD)是真核生物的基因组三维结构的基本组成单位。然而,最近的研究表明位于哺乳动物TAD边界的绝缘复合物CTCF /cohesin在黑腹果蝇中是缺失的,暗示着边界组成元素在后生动物中的不保守性。本文采用了亚Kb分辨率的原位Hi-C,发现果蝇基因组几乎完全被划分到> 4000 个TAD中,比以前确定的多7倍以上。绝大多数TAD边界的绝缘复合物为BEAF-32/CP190,或者BEAF-32/Chromator,表明这些蛋白质也许跟CTCF /cohesin一样在哺乳动物中起类似的作用。新找到的TAD之前被认为是没有结构的,但在本文发现是一些小的连续的TAD,这在人的基因组中也观察到了。总之,本文揭示了黑腹果蝇染色体基本的结构特征及其结构在昆虫到哺乳动物中的保守性(以后大方向,做基因组不光需要组装序列,三维结构解析也是少不了的,我们可以全方位提供哦~)。
1. 分辨率提高到200bp获得大量新的小的TAD
图1-a为3号染色体左臂3.5-6.3Mb区间热图。左边的热图为20Kb的分辨率,显示清晰的Super-TAD(热图下方蓝色对应区域)和Super-TAD之间区域(热图底部红色对应区域),与以前的研究结果相一致(Super-TAD表示之前研究由于分辨率较低而鉴定的普遍较大的TAD)。但在更高的分辨率(~ 200 bp),热图显示Super-TAD(图1-a右上热图)和Super-TAD之间(图1-a右下方热图)均发现许多连续的小TAD,热图下方红色对应的区域代表新鉴定的TAD。
图1-a为TAD长度分布图,总共鉴定到了4,123 TADs,长度范围3-460Kb, 平均大小13Kb,覆盖>92%的基因组的非重复区。
图1-b展示了TAD在Super-TAD内部(蓝色)和Super-TAD之间(红色)的数量分布。
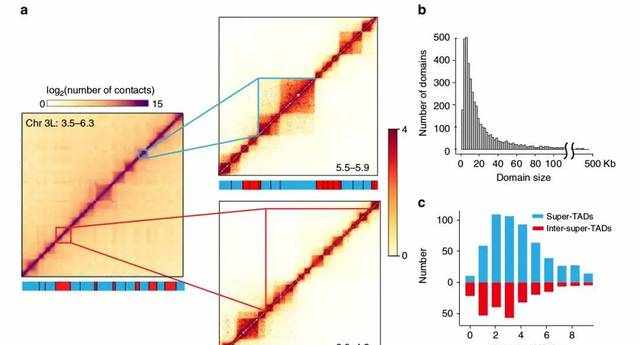
图1 果蝇TAD分布图
2. 果蝇TAD边界富集有绝缘蛋白,但并非CTCF
图2-a显示I类绝缘子蛋白在TAD边界富集。具体到某一类绝缘子蛋白,可以看到beaf-32,cp190和Chromator在TAD边界富集。图2-a最下面一行显示的是成对的绝缘体蛋白BEAF-32/CP190和BEAF-32/Chromator分布在TAD边界的位置。
图2-b表示全基因组范围内绝缘体蛋白与成对绝缘体蛋白的位置与TAD边界的重叠程度。超过>91%的TAD边界包含三种绝缘蛋白中的一个。

图2 TAD边界与绝缘蛋白的分布
3. 表观遗传修饰语TAD之间的交互关系
根据15个组蛋白修饰标记和非组蛋白的染色体蛋白在TAD内部的分布可以将一个TAD分为四个主要类型(图3-a):活性(橙色),失活(蓝色),Polycomb(绿色)和不确定(灰色)。
图3-a展示了活性TAD和失活TAD与直接相邻的TAD的交互作用强弱比较。其中上图是原始交互热图,下图是交互值相对于期望的交互值的的热图。红色框为活性的TAD,邻近TAD交互较弱,蓝色框为失活的TAD,邻近TAD交互较强。
图3-b三种类型TAD之间相对作用强度显示inactive-inactive和polycomb-polycomb TAD 之间交互信号强,active-active TAD 之间交互信号弱。
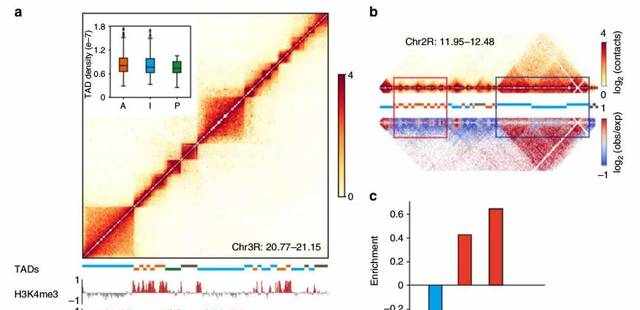
图3 不同类型TAD之间的交互差异
4. 果蝇TAD与哺乳动物TAD的保守性
人的基因组在高分辨率(5Kb)条件下TAD之间又重新鉴定出连续的小的TAD,其中红色条形区域为新鉴定的TAD。同时此图也展示了CTCF (橘色) 和cohesin components (Rad21为绿色箭头,Smc3棕色箭头)在TAD边界的分布。因此除了绝缘蛋白种类不一样,果蝇TAD结构与TAD边界分布有绝缘蛋白等特征与哺乳动物都是一致的。

图4 高分辨的人基因组的TAD热图(5Kb)
参考文献:
Wang, Q., Sun, Q., Czajkowsky, D. M., & Shao, Z. (2018). Sub-kb hi-c in d.melanogaster reveals conserved characteristics of tads between insect and mammalian cells. Nature Communications, 9(1), 188.
1.《NC—果蝇基因组染色体三维结构解析》援引自互联网,旨在传递更多网络信息知识,仅代表作者本人观点,与本网站无关,侵删请联系页脚下方联系方式。
2.《NC—果蝇基因组染色体三维结构解析》仅供读者参考,本网站未对该内容进行证实,对其原创性、真实性、完整性、及时性不作任何保证。
3.文章转载时请保留本站内容来源地址,https://www.lu-xu.com/shehui/21853.html