抽象的
为建立黄曲霉毒素B1的灵敏免疫分析方法,筛选了一株分泌高亲和力抗黄曲霉毒素B1单克隆抗体的杂交瘤细胞。合成了一种新型碲化镉/硫化镉/硫化锌量子点,并将其与人工抗原偶联,作为荧光探针用于简单的一步荧光免疫分析。所研制的荧光分析仪可在0.08 ~ 1.97纳克/毫升的宽线性范围内灵敏地测定粮食样品中的黄曲霉毒素B1,粮食样品的检出限为0.01纳克/毫升。克隆并测序了Fab片段的免疫球蛋白基因,成功验证了Fab在HEK293细胞中的表达。黄曲霉毒素B1的分子量为1.09×10-7摩尔/升..
为了研究抗体与AFB1的相互作用,采用分子对接和分子动力学方法模拟了AFB1和Fab片段的同源模型。结果表明,Ser32、Trp93和Trp98残基在氢键、π-π堆积/π-烷基相互作用和范德华相互作用中起着最重要的作用。此外,对AFB1的静电势研究表明,静电相互作用在识别过程中也起着重要作用。理论结果为半抗原的设计和基因工程抗体的改良提供了指导。
同源性建模是利用信息技术的一种手段,可以从一级结构(氨基酸序列)直接预测蛋白质的高级结构(主要是三级结构)。人们可以利用一种或多种已知的结构蛋白(模板)来构建未知结构蛋白(靶蛋白)的空结构。Discovery Studio通过使用同源建模的方法,为用户提供了一套自动预测蛋白质之间空结构的工具。用户只需提供蛋白质的氨基酸序列,即可轻松完成同源模型的构建和模型可信度的评估。
分子对接是基于结构药物设计的核心模拟方法,根据受体-配体相互作用的几何匹配和能量匹配过程模拟受体-配体相互作用,预测二者之间的最佳结合方式和结合亲和力。利用分子对接模拟技术,研究人员可以基于结构对药物进行虚拟筛选,对药物分子进行结构修饰,研究配体-受体相互作用的机制,从而大大提高实验效率。在分子模拟综合平台Discovery Studio中,分子对接程序包括Libdock、CD locker和Flexible Docking。这三种对接算法各有优势,既能满足研究者的各种应用需求,又为他们提供了配体与受体之间相互识别的“利器”。
◆ ◆ ◆ ◆
碲化镉/硫化镉/硫化锌量子点
黄曲霉毒素B1的免疫测定和抗体识别
分子模型中的应用
参考:中国分析学报。2018年9月26日在线提供,中频=5.256
链接:https://doi.org/10.1016/j.aca.2018.09.058
◆ ◆ ◆ ◆
研究背景
B1(AFB1)是一种真菌毒素,主要由寄生曲霉和黄曲霉在谷物成熟、收获和储藏过程中,在各种环境条件下产生。黄曲霉毒素B1污染食物链和环境,会对人类、家禽和家畜产生各种潜在的有害后果。如致癌、胚胎毒性、免疫抑制、致畸等。免疫分析因其适用性、灵敏度和快速反应性,已成为短时间内分析大量样品的最常用方法。然而,荧光免疫分析法比传统免疫分析法更灵敏,因此其利用率在不断提高。同时,由于其独特的光谱特性,镉量子点作为荧光信号探针的使用也有所增加。如CdTe/CdS/ZnS量子点含有CdS和ZnS的壳层,不仅可以急剧增加荧光强度,还可以阻止有毒Cd2+的释放,可以作为荧光探针,在细胞标记和靶标分析中具有广阔的前景。
Fab的克隆和测序
用聚合酶链反应从抗AFB1单克隆杂交瘤细胞中扩增出Fd和LC基因,扩增产物分别为660 bp和642 bp扩增子。该基因由卡巴特数据库和发现工作室2016测序。结果表明,扩增的VL基因属于IGV1基因家族,而VH序列属于IGV1基因家族。基因序列显示无终止密码子的框内连接,表明序列重组正确,氨基酸序列相关。
单克隆抗体的Fab片段建模
利用Discovery Studio 2016软件构建了mAb 5A11C10A Fab片段的三维结构。轻链和重链的模板结构首先在PDB数据库中通过BLAST搜索来识别。为了建模的准确性,分别选择1MFMB、3BSZ和3KS0的抗体晶体结构作为整体、重链和轻链模板。然后通过在整个模板上叠加重链和轻链的模板来确定两条链的相对空方向,从而构建Fab片段的结构。Ramachandran图和Profile-3D分析用于验证Fab蛋白中预测的扭转角。
分子对接和分子动力学模拟
为了研究AFB1抗体识别的特异性结合机制,作者在Discovery Studio中使用了半柔性分子对接方法CD Locker。结果表明,AFB1非常适合CDR区形成的疏水腔,说明基于几何匹配原理,AFB1可以有效匹配Fab的活性位点,CDR区的残基在AFB1抗体的识别中起着重要作用。氢键和π-π堆积/π-烷基相互作用是结合AFB1和Fab的关键相互作用。为了获得更好的模拟结果,利用分子动力学模拟了最佳对接模型,计算了AFB1和Fab的结合自由能。
结论
在本研究中,作者制备了一种新的抗黄曲霉毒素B1单克隆抗体,并合成了一种相对新的CdTe/CdS/ZnS三元量子点,可用于黄曲霉毒素B1的灵敏免疫分析,该三元量子点可作为荧光探针。与传统的酶联免疫吸附试验相比,该方法的检测限降低了4倍。然后,根据同源性建模方法,构建Fab片段的三维同源性模型,进行分子对接和分子动力学模拟,研究AFB1抗体的识别机制。这些方法可以帮助我们更好地理解半抗原抗体的识别机制,指导未来半抗原的设计,通过基因工程提高抗体。
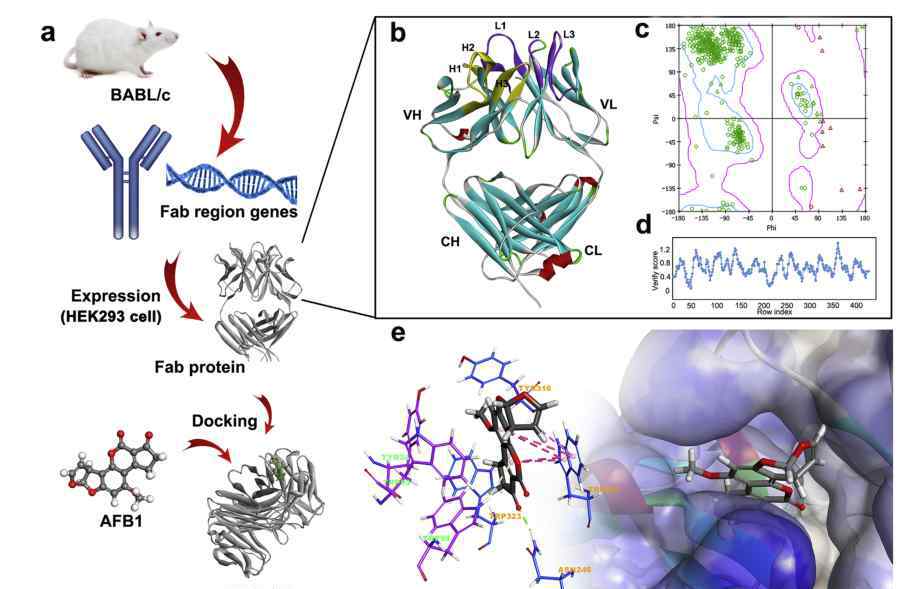
图1 (a) Fab表达和分子建模工作流程;(二)Fab三维模型和互补决定区;(c) Ramachandran图和(fab域模型的剖面-3D分析;(e) Fab结合位点以及对接后Fab与AFB1的相互作用。
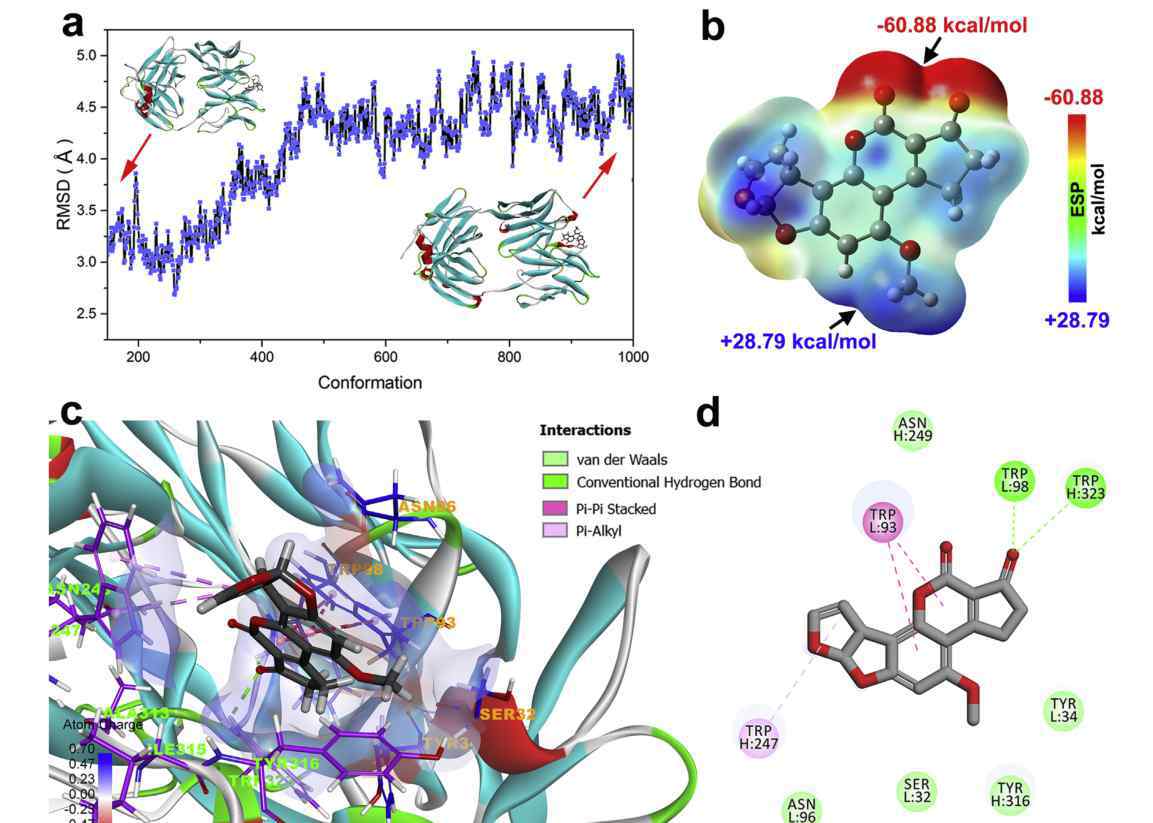 图2 分子对接与分子动力学模拟的结果图图2分子对接和分子动力学模拟结果
图2 分子对接与分子动力学模拟的结果图图2分子对接和分子动力学模拟结果
 表1 Fab-AFB1复合物中每个残基的相互作用能的分解值表1 FAB-AFB 1复合物中各残基相互作用能的分解值
表1 Fab-AFB1复合物中每个残基的相互作用能的分解值表1 FAB-AFB 1复合物中各残基相互作用能的分解值
1.《afb1 案例|CdTe /CdS /ZnS量子点在黄曲霉毒素B1免疫分析及抗体识别分子模型中的应》援引自互联网,旨在传递更多网络信息知识,仅代表作者本人观点,与本网站无关,侵删请联系页脚下方联系方式。
2.《afb1 案例|CdTe /CdS /ZnS量子点在黄曲霉毒素B1免疫分析及抗体识别分子模型中的应》仅供读者参考,本网站未对该内容进行证实,对其原创性、真实性、完整性、及时性不作任何保证。
3.文章转载时请保留本站内容来源地址,https://www.lu-xu.com/fangchan/1256149.html
